Apple dévoile SimpleFold, une IA « légère » et prédictive pour les protéines
C’est une sacré surprise : se plaçant en concurrent direct de l’AlphaFold de Google DeepMind, les chercheurs d’Apple viennent de présenter SimpleFold, un modèle d’intelligence artificielle conçu pour prédire la structure tridimensionnelle des protéines tout en réduisant considérablement les coûts de calcul. Un défi de taille quand on sait que cette tâche nécessitait auparavant des mois de travail contre seulement quelques heures aujourd’hui grâce à ces nouveaux modèles de pointe.
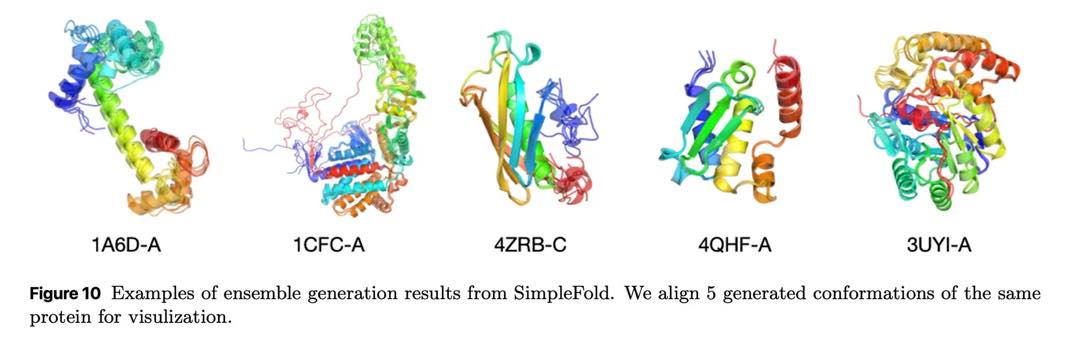
Un modèle plus simple et moins coûteux
Contrairement à AlphaFold2 ou RoseTTAFold, qui reposent sur des architectures complexes intégrant alignements de séquences multiples et mises à jour géométriques sophistiquées, SimpleFold s’appuie sur les modèles de flow matching. Popularisée en 2023 dans le domaine de l’image et de la 3D, cette approche permet de générer des résultats en suivant un chemin plus direct, sans les étapes lourdes de débruitage. Résultat : une efficacité accrue, aussi bien en entraînement qu’en inférence.
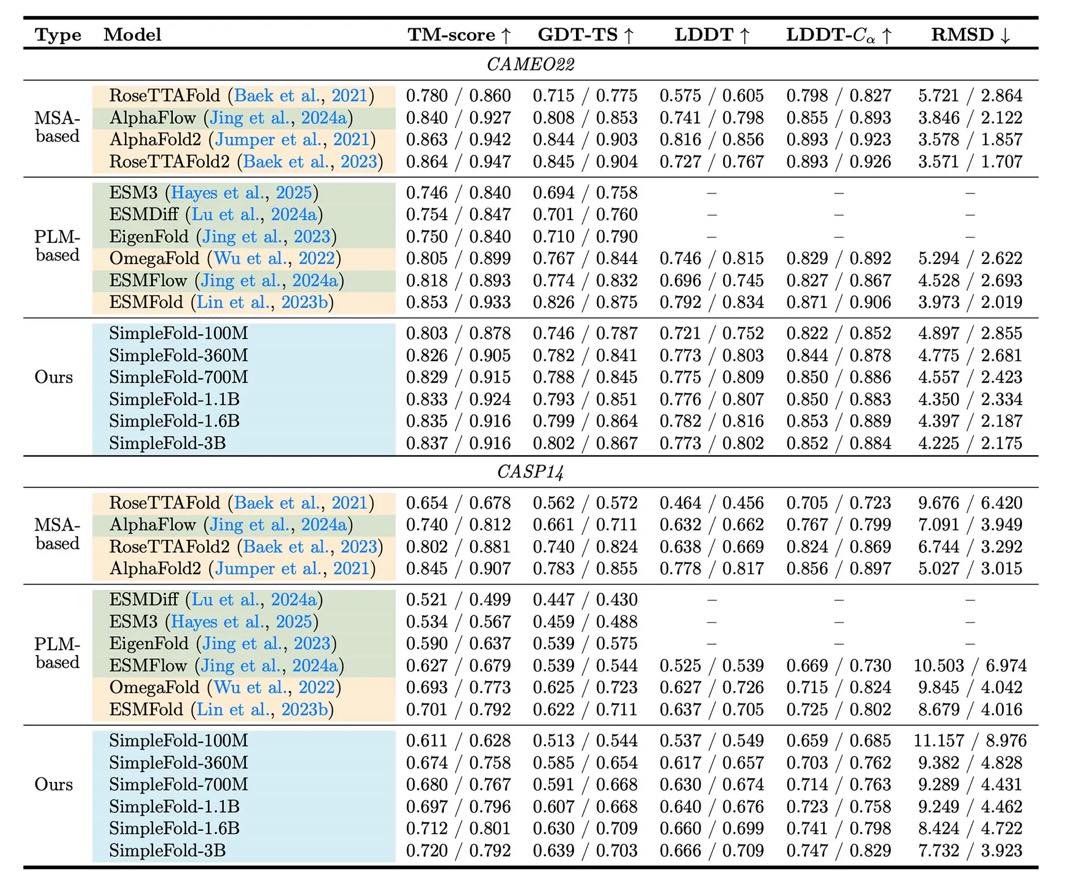
Des performances déjà prometteuses
Testé sur les benchmarks CAMEO22 et CASP14, SimpleFold a obtenu des scores comparables aux meilleurs modèles du marché, atteignant plus de 95 % des performances d’AlphaFold2 et RoseTTAFold2 sur plusieurs critères. Même sa version la plus compacte, dotée d' »à peine » 100 millions de paramètres, rivalise efficacement avec ESMFold, tout en restant bien plus légère. « Malgré sa simplicité, SimpleFold démontre des performances compétitives », notent ainsi les chercheurs.
Pour Apple, ce projet n’est qu’une première étape. L’équipe espère en effet que cette innovation incitera la communauté scientifique à développer des modèles génératifs de protéines toujours plus puissants et économes, ouvrant de nouvelles perspectives pour la biologie et la médecine du futur. Reste encore à savoir comment cette avancée pourra s’intégrer dans de futurs services ou appareils de santé d’Apple, voire servir à la recherche médicale.


Après, on dira que Apple est mort et ne s’inove plus 🤣🤣